This site is not complete. The work to converting the volumes of സര്വ്വവിജ്ഞാനകോശം is on progress. Please bear with us
Please contact webmastersiep@yahoo.com for any queries regarding this website.
Reading Problems? see Enabling Malayalam
ജീന് മാപ്പിങ്
സര്വ്വവിജ്ഞാനകോശം സംരംഭത്തില് നിന്ന്
(പുതിയ താള്: ==ജീന് മാപ്പിങ്== ==Gene mapping== ഒരു ജീവിയുടെ ജീനുകള് അതിന്റെ ക്രോമസോ...) |
(→Gene mapping) |
||
| വരി 14: | വരി 14: | ||
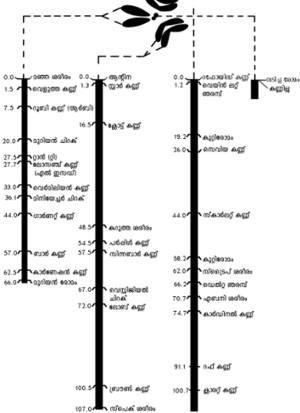
'''ത്രിബിന്ദു ടെസ്റ്റ് ക്രോസ്.''' രണ്ടിലധികം ജീനുകളെ മാപ്പ് ചെയ്യേണ്ടിവരുമ്പോള് ഈ രീതിയാണു കൂടുതല് ഫലപ്രദം. അടുത്തടുത്ത മൂന്നു ജീനുകള്ക്കിടയ്ക്ക് ഒരേ സമയം രണ്ടു ജീന് വിനിമയം നടക്കുന്നതിനെ ഇരട്ട ക്രോസ് ഓവര് (നോ. ജീന് വിനിമയം) എന്നു പറയുന്നു. അതു കണ്ടെത്താനും അതിന്റെ ശ.കൂടി മാപ്പില് ഉള്പ്പെടുത്താനും ത്രിബിന്ദു ടെസ്റ്റ് ക്രോസ് കൊണ്ടേ കഴിയുകയുള്ളു. രണ്ടു ജീനുകള് തമ്മിലുള്ള പരമാവധി ക്രോസിങ് ഓവര് 50% ആണ്. ഇത് 50 മാപ്പ് യൂണിറ്റിനു തുല്യമാണ്. എന്നാല് ഇരട്ട ജീന് വിനിമയ മൂല്യം ആവര്ത്തിച്ച് ഓരോ ജോടി ജീനുകളുടെ ഒപ്പവും കൂട്ടുന്നതുകൊണ്ട് ചില വലിയ ക്രോമസോമുകളുടെ രണ്ടറ്റത്തുമുള്ള ജീനുകള് തമ്മില് നൂറു മാപ്പ് യൂണിറ്റോ അതിലധികമോ അകലം കണ്ടുവരുന്നു. ഡ്രോസോഫില മെലനോഗാസ്റ്റര് എന്ന പഴ ഈച്ചയുടെ നാലു ജോടി ക്രോമസോമുകളുടെ ജീന് മാപ്പ് ചിത്രത്തില് കാണിച്ചിരിക്കുന്നു: | '''ത്രിബിന്ദു ടെസ്റ്റ് ക്രോസ്.''' രണ്ടിലധികം ജീനുകളെ മാപ്പ് ചെയ്യേണ്ടിവരുമ്പോള് ഈ രീതിയാണു കൂടുതല് ഫലപ്രദം. അടുത്തടുത്ത മൂന്നു ജീനുകള്ക്കിടയ്ക്ക് ഒരേ സമയം രണ്ടു ജീന് വിനിമയം നടക്കുന്നതിനെ ഇരട്ട ക്രോസ് ഓവര് (നോ. ജീന് വിനിമയം) എന്നു പറയുന്നു. അതു കണ്ടെത്താനും അതിന്റെ ശ.കൂടി മാപ്പില് ഉള്പ്പെടുത്താനും ത്രിബിന്ദു ടെസ്റ്റ് ക്രോസ് കൊണ്ടേ കഴിയുകയുള്ളു. രണ്ടു ജീനുകള് തമ്മിലുള്ള പരമാവധി ക്രോസിങ് ഓവര് 50% ആണ്. ഇത് 50 മാപ്പ് യൂണിറ്റിനു തുല്യമാണ്. എന്നാല് ഇരട്ട ജീന് വിനിമയ മൂല്യം ആവര്ത്തിച്ച് ഓരോ ജോടി ജീനുകളുടെ ഒപ്പവും കൂട്ടുന്നതുകൊണ്ട് ചില വലിയ ക്രോമസോമുകളുടെ രണ്ടറ്റത്തുമുള്ള ജീനുകള് തമ്മില് നൂറു മാപ്പ് യൂണിറ്റോ അതിലധികമോ അകലം കണ്ടുവരുന്നു. ഡ്രോസോഫില മെലനോഗാസ്റ്റര് എന്ന പഴ ഈച്ചയുടെ നാലു ജോടി ക്രോമസോമുകളുടെ ജീന് മാപ്പ് ചിത്രത്തില് കാണിച്ചിരിക്കുന്നു: | ||
| + | |||
| + | [[ചിത്രം:Pg 737 jeen mapingsr.png|300px]] | ||
ടെസ്റ്റ് ക്രോസില് നിന്നല്ലാതെ ഒരു ക്രോസിന്റെ രണ്ടാം തലമുറയിലെ (F<sub>2</sub>) വിവരത്തില് നിന്നും ജീന് വിനിമയ മാപ്പ് നിര്മിക്കാം. ഒരു ജീന് വിനിമയ മാപ്പ് ജീനുകളുടെ ആപേക്ഷിക അകലത്തിന്റെ സൂചനയേ നല്കുന്നുള്ളു. ക്രോമസോമില് ജീനിന്റെ യഥാര്ഥ സ്ഥാനം നിര്ണയിക്കാന് സൈറ്റോജനറ്റിക് മാപ്പുകള് (cytogenetic maps) ഉപയോഗപ്പെടുത്താം. | ടെസ്റ്റ് ക്രോസില് നിന്നല്ലാതെ ഒരു ക്രോസിന്റെ രണ്ടാം തലമുറയിലെ (F<sub>2</sub>) വിവരത്തില് നിന്നും ജീന് വിനിമയ മാപ്പ് നിര്മിക്കാം. ഒരു ജീന് വിനിമയ മാപ്പ് ജീനുകളുടെ ആപേക്ഷിക അകലത്തിന്റെ സൂചനയേ നല്കുന്നുള്ളു. ക്രോമസോമില് ജീനിന്റെ യഥാര്ഥ സ്ഥാനം നിര്ണയിക്കാന് സൈറ്റോജനറ്റിക് മാപ്പുകള് (cytogenetic maps) ഉപയോഗപ്പെടുത്താം. | ||
| വരി 22: | വരി 24: | ||
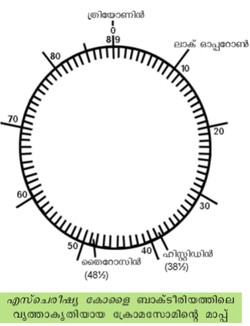
'''കോണ്ജുഗേഷന് മാപ്പിങ് (conjugation mapping)'''. രണ്ടു ബാക്റ്റീരിയങ്ങള് പരസ്പരം ബന്ധപ്പെട്ട് അതില് ഒന്നില് നിന്നു മറ്റേതിലേക്കു മാത്രം നടക്കുന്ന ജീന് കൈമാറ്റമാണ് കോണ്ജുഗേഷന്. കോണ്ജുഗേഷന്റെ സമയം കൂടുന്തോറും കടന്നുപോകുന്ന ജീനുകളുടെ എണ്ണവും കൂടും. ഒരു ബാക്റ്റീരിയത്തിന്റെ ക്രോമസോം മുറിഞ്ഞിട്ട് അതിലെ ഒരു ഡി.എന്.എ. ഇഴയാണ് വേറൊരു ബാക്റ്റീരിയത്തിലേക്കു കടക്കുന്നത്. ക്രോമസോം മുറിയുന്ന സ്ഥലവും ആദ്യം കടക്കുന്ന ജീനും ഏറെക്കുറെ സ്ഥിരമാണ്. അപ്പോള് ആദ്യ ജീനിനു ശേഷം കടക്കുന്ന ഓരോ ജീനും ഒരു പ്രത്യേക കാലയളവിലായിരിക്കും കടന്നുപോവുക. കോണ്ജുഗേഷനെ വ്യത്യസ്ത കാലയളവുകളില് തടസ്സപ്പെടുത്തിയാല് ഓരോ കാലയളവിലും കടന്നുപോകുന്ന ജീനിനെ കണ്ടെത്താം. ജേക്കബ്, വുള്മാന് എന്നിവരാണ് ഈ രീതി ആവിഷ്കരിച്ചത്. ഇങ്ങനെ നിര്മിച്ച എസ്ചെരിഷ്യ കോളൈ ബാക്റ്റീരിയത്തിലെ വൃത്താകൃതിയായ ക്രോമസോമിന്റെ ഒരു മാപ്പ് ചിത്രത്തില് ചേര്ത്തിരിക്കുന്നു: | '''കോണ്ജുഗേഷന് മാപ്പിങ് (conjugation mapping)'''. രണ്ടു ബാക്റ്റീരിയങ്ങള് പരസ്പരം ബന്ധപ്പെട്ട് അതില് ഒന്നില് നിന്നു മറ്റേതിലേക്കു മാത്രം നടക്കുന്ന ജീന് കൈമാറ്റമാണ് കോണ്ജുഗേഷന്. കോണ്ജുഗേഷന്റെ സമയം കൂടുന്തോറും കടന്നുപോകുന്ന ജീനുകളുടെ എണ്ണവും കൂടും. ഒരു ബാക്റ്റീരിയത്തിന്റെ ക്രോമസോം മുറിഞ്ഞിട്ട് അതിലെ ഒരു ഡി.എന്.എ. ഇഴയാണ് വേറൊരു ബാക്റ്റീരിയത്തിലേക്കു കടക്കുന്നത്. ക്രോമസോം മുറിയുന്ന സ്ഥലവും ആദ്യം കടക്കുന്ന ജീനും ഏറെക്കുറെ സ്ഥിരമാണ്. അപ്പോള് ആദ്യ ജീനിനു ശേഷം കടക്കുന്ന ഓരോ ജീനും ഒരു പ്രത്യേക കാലയളവിലായിരിക്കും കടന്നുപോവുക. കോണ്ജുഗേഷനെ വ്യത്യസ്ത കാലയളവുകളില് തടസ്സപ്പെടുത്തിയാല് ഓരോ കാലയളവിലും കടന്നുപോകുന്ന ജീനിനെ കണ്ടെത്താം. ജേക്കബ്, വുള്മാന് എന്നിവരാണ് ഈ രീതി ആവിഷ്കരിച്ചത്. ഇങ്ങനെ നിര്മിച്ച എസ്ചെരിഷ്യ കോളൈ ബാക്റ്റീരിയത്തിലെ വൃത്താകൃതിയായ ക്രോമസോമിന്റെ ഒരു മാപ്പ് ചിത്രത്തില് ചേര്ത്തിരിക്കുന്നു: | ||
| + | |||
| + | [[ചിത്രം:Jeen mapping 699scrt.png|250px]] | ||
'''ബാക്റ്റീരിയ ജീന് മാപ്പിങ്ങിന്റെ മറ്റു മാര്ഗങ്ങള്.''' കോണ്ജുഗേഷനു പുറമേ ട്രാന്സ്ഡക്ഷന്, ട്രാന്സ്ഫൊര്മേഷന് എന്നീ മാര്ഗങ്ങളിലൂടെയും ബാക്റ്റീരിയങ്ങളില് ജീന് വിനിമയം നടക്കാം. | '''ബാക്റ്റീരിയ ജീന് മാപ്പിങ്ങിന്റെ മറ്റു മാര്ഗങ്ങള്.''' കോണ്ജുഗേഷനു പുറമേ ട്രാന്സ്ഡക്ഷന്, ട്രാന്സ്ഫൊര്മേഷന് എന്നീ മാര്ഗങ്ങളിലൂടെയും ബാക്റ്റീരിയങ്ങളില് ജീന് വിനിമയം നടക്കാം. | ||
Current revision as of 07:27, 24 ഫെബ്രുവരി 2016
ജീന് മാപ്പിങ്
Gene mapping
ഒരു ജീവിയുടെ ജീനുകള് അതിന്റെ ക്രോമസോമില് കാണുന്നതിന്റെ ക്രമവും അകലവും കണ്ടെത്തല്. ക്രോമസോമിലാണ് ജീനുകള് എന്ന സിദ്ധാന്തം ഡബ്ല്യു.എസ്. സട്ടണും റ്റി. ബോവറിയും നിര്ദേശിച്ചു (1902). ക്രോമസോം വഴിയുള്ള ജീനിന്റെ പാരമ്പര്യം റ്റി.എച്ച്. മോര്ഗന് വ്യക്തമാക്കി (1910). ഒരേ ക്രോമസോമിലുള്ള ഒന്നിലധികം ജീനുകള് പാരമ്പര്യത്തില് ഒന്നിച്ചിരിക്കാന് കാട്ടുന്ന വാസനയെ ലിങ്കേജ് (Linkage) എന്നു മോര്ഗന് നാമകരണം ചെയ്തു. ജീനുകള് ക്രോമസോമില് ഒരു നിരയാ(Linear arrangement)യാണ് കാണുന്നതെന്നു മോര്ഗന് കണ്ടെത്തി (നോ. ജീന്, ക്രോമസോം). ഒരേ ക്രോമസോമില് അടുത്തു കാണുന്ന ജീനുകള് തമ്മില് ജീന് വിനിമയം (നോ. ജീന് വിനിമയം) കുറവാണെന്നും ക്രോമസോമില് രണ്ടു ജീനുകള് അകലെയാണെങ്കില് അവയ്ക്കിടയില് ജീന് വിനിമയം കൂടുതലായിരിക്കുമെന്നും നിരീക്ഷിക്കപ്പെട്ടു. അങ്ങനെയാണെങ്കില് ജീന് വിനിമയത്തിന്റെ ശതമാനത്തെ (percentage of crossing over) ജീനുകള് തമ്മിലുള്ള അകലത്തിന്റെ അളവുകോലായി കണക്കാക്കാം. ഈ തത്ത്വത്തെ അടിസ്ഥാനമാക്കി എ.എച്ച്. സ്റ്റര്ട്ടിവാന്റ് ജീന് മാപ്പിങ്ങിനുള്ള മാര്ഗം ആവിഷ്കരിച്ചു (1913). ഒരു ശ. ജീന് വിനിമയം ജീനുകള് തമ്മില് ഒരു യൂണിറ്റ് അകലം സൂചിപ്പിക്കുന്നതായി ഇദ്ദേഹം കണക്കാക്കി. ഈ മാപ്പ് യൂണിറ്റിനു സെന്റി മോര്ഗന് എന്ന പേരും നല്കി.
ജീന് വിനിമയ ശ. കണക്കാക്കി ജീന് മാപ്പു ചെയ്യുന്നതിനു ദ്വിഗുണിത ജീവികളില് (ക്രോമസോമുകള് ജോടിയായിക്കാണുന്ന ജീവികള്) ദ്വി ബിന്ദു ടെസ്റ്റ് ക്രോസ് (Two Point test cross), ത്രി ബിന്ദു ടെസ്റ്റ് ക്രോസ് (Three-point test cross) എന്ന രണ്ടു മാര്ഗങ്ങളാണു സാധാരണ ഉപയോഗിക്കുന്നത്.
ദ്വിബിന്ദു ടെസ്റ്റ് ക്രോസ്. രണ്ടു ജീനുകള് തമ്മിലുള്ള അകലം കണ്ടെത്താന് ഏറ്റവും ലളിതമായ മാര്ഗമാണിത്. രണ്ടു ജീനുകള് ഉള്പ്പെടുന്ന ഈ ടെസ്റ്റ് ക്രോസ് താഴെക്കാണിച്ചിരിക്കുന്നു:
മുകളില് കാണിച്ച ഉദാഹരണത്തില് ക്രോസ് ഓവര് ഇനം കുട്ടികള് 20% ആണ്. 20% ജീന് വിനിമയം a യും b യും ജീനുകള്ക്കിടയില് നടന്നു എന്നതിന്റെ സൂചനയാണിത്. ഒരു ശ.ജീന് വിനിമയം ഒരു മാപ്പ് യൂണിറ്റ് എന്ന കണക്കില് ജീന് a യും b യും തമ്മിലുള്ള അകലം 20 മാപ്പ് യൂണിറ്റ്. ഈ രണ്ടു ജീനുകളുടെ മാപ്പ് താഴെ കാണിച്ചിരിക്കുന്നു.
ത്രിബിന്ദു ടെസ്റ്റ് ക്രോസ്. രണ്ടിലധികം ജീനുകളെ മാപ്പ് ചെയ്യേണ്ടിവരുമ്പോള് ഈ രീതിയാണു കൂടുതല് ഫലപ്രദം. അടുത്തടുത്ത മൂന്നു ജീനുകള്ക്കിടയ്ക്ക് ഒരേ സമയം രണ്ടു ജീന് വിനിമയം നടക്കുന്നതിനെ ഇരട്ട ക്രോസ് ഓവര് (നോ. ജീന് വിനിമയം) എന്നു പറയുന്നു. അതു കണ്ടെത്താനും അതിന്റെ ശ.കൂടി മാപ്പില് ഉള്പ്പെടുത്താനും ത്രിബിന്ദു ടെസ്റ്റ് ക്രോസ് കൊണ്ടേ കഴിയുകയുള്ളു. രണ്ടു ജീനുകള് തമ്മിലുള്ള പരമാവധി ക്രോസിങ് ഓവര് 50% ആണ്. ഇത് 50 മാപ്പ് യൂണിറ്റിനു തുല്യമാണ്. എന്നാല് ഇരട്ട ജീന് വിനിമയ മൂല്യം ആവര്ത്തിച്ച് ഓരോ ജോടി ജീനുകളുടെ ഒപ്പവും കൂട്ടുന്നതുകൊണ്ട് ചില വലിയ ക്രോമസോമുകളുടെ രണ്ടറ്റത്തുമുള്ള ജീനുകള് തമ്മില് നൂറു മാപ്പ് യൂണിറ്റോ അതിലധികമോ അകലം കണ്ടുവരുന്നു. ഡ്രോസോഫില മെലനോഗാസ്റ്റര് എന്ന പഴ ഈച്ചയുടെ നാലു ജോടി ക്രോമസോമുകളുടെ ജീന് മാപ്പ് ചിത്രത്തില് കാണിച്ചിരിക്കുന്നു:
ടെസ്റ്റ് ക്രോസില് നിന്നല്ലാതെ ഒരു ക്രോസിന്റെ രണ്ടാം തലമുറയിലെ (F2) വിവരത്തില് നിന്നും ജീന് വിനിമയ മാപ്പ് നിര്മിക്കാം. ഒരു ജീന് വിനിമയ മാപ്പ് ജീനുകളുടെ ആപേക്ഷിക അകലത്തിന്റെ സൂചനയേ നല്കുന്നുള്ളു. ക്രോമസോമില് ജീനിന്റെ യഥാര്ഥ സ്ഥാനം നിര്ണയിക്കാന് സൈറ്റോജനറ്റിക് മാപ്പുകള് (cytogenetic maps) ഉപയോഗപ്പെടുത്താം.
സൈറ്റോജനറ്റിക് മാപ്പ്. മൈക്രോസ്കോപ്പ് ഉപയോഗിച്ചുള്ള ക്രോമസോം പഠനത്തിലൂടെ ജീനുകളുടെ സ്ഥാനം നിശ്ചയിച്ചാണ് ഇത്തരം മാപ്പുണ്ടാക്കുക. ജീനുകളെ മൈക്രോസ്കോപ്പിലൂടെ കാണാനോ തിരിച്ചറിയാനോ സാധ്യമല്ല (ഇലക്ട്രോണ് മൈക്രോസ്കോപ്പിലൂടെ ഡി.എന്.എ. തന്മാത്ര കാണാമെങ്കിലും സാധാരണ മൈക്രോസ്കോപ്പുകളാണ് സൈറ്റോജനറ്റിക് മാപ്പുണ്ടാക്കാന് ഉപയോഗിച്ചിരുന്നത്). ക്രോമസോം ഖണ്ഡങ്ങളുടെ നഷ്ടങ്ങള് (deletions), ഇരട്ടിക്കലുകള് (duplications) തുടങ്ങിയ വൈകല്യങ്ങള് (നോ. ജനിതക വൈകല്യങ്ങള്) ജീവിയുടെ സ്വഭാവങ്ങളെ എങ്ങനെ ബാധിക്കുന്നു എന്നു നിരീക്ഷിക്കുന്നു. ചില സ്വഭാവങ്ങളുടെ ജീനുകള് ചില പ്രത്യേക ക്രോമസോം ഖണ്ഡങ്ങളുമായി ബന്ധപ്പെട്ടിരിക്കുന്നതായി ഇതിലൂടെ മനസ്സിലാക്കാം. ഡ്രോസോഫിലയുടെ സാധാരണ ക്രോമോസോമുകളില് എച്ച്.ജെ. മുള്ളര് തുടങ്ങിവച്ച ഇത്തരം ജീന് മാപ്പിങ് ഡ്രോസോഫിലയുടെ തന്നെ ഉമിനീര് ഗ്രന്ഥികളിലെ ഭീമ ക്രോമസോമുകള് (നോ. ക്രോമസോം) ഉപയോഗിച്ച് റ്റി.എച്ച്. പെയ്ന്റര് കൂടുതല് ഫലവത്താക്കി. തുടര്ന്ന് 1930-കളില് ബ്രിഡ്ജസ് നടത്തിയ പഠനങ്ങള് ഡ്രോസോഫിലയുടെ വിശദമായ സെറ്റോളജിക്കല് മാപ്പുകളുടെ നിര്മാണത്തിനു വഴിയൊരുക്കി.
ബാക്റ്റീരിയങ്ങളിലെ ജീന് മാപ്പിങ്. ഒറ്റ ക്രോമസോം മാത്രമുള്ള ബാക്റ്റീരിയങ്ങളിലെ ജീനുകളെ - വിവിധ മാര്ഗങ്ങളിലൂടെ മാപ്പ് ചെയ്യാവുന്നതാണ്.
കോണ്ജുഗേഷന് മാപ്പിങ് (conjugation mapping). രണ്ടു ബാക്റ്റീരിയങ്ങള് പരസ്പരം ബന്ധപ്പെട്ട് അതില് ഒന്നില് നിന്നു മറ്റേതിലേക്കു മാത്രം നടക്കുന്ന ജീന് കൈമാറ്റമാണ് കോണ്ജുഗേഷന്. കോണ്ജുഗേഷന്റെ സമയം കൂടുന്തോറും കടന്നുപോകുന്ന ജീനുകളുടെ എണ്ണവും കൂടും. ഒരു ബാക്റ്റീരിയത്തിന്റെ ക്രോമസോം മുറിഞ്ഞിട്ട് അതിലെ ഒരു ഡി.എന്.എ. ഇഴയാണ് വേറൊരു ബാക്റ്റീരിയത്തിലേക്കു കടക്കുന്നത്. ക്രോമസോം മുറിയുന്ന സ്ഥലവും ആദ്യം കടക്കുന്ന ജീനും ഏറെക്കുറെ സ്ഥിരമാണ്. അപ്പോള് ആദ്യ ജീനിനു ശേഷം കടക്കുന്ന ഓരോ ജീനും ഒരു പ്രത്യേക കാലയളവിലായിരിക്കും കടന്നുപോവുക. കോണ്ജുഗേഷനെ വ്യത്യസ്ത കാലയളവുകളില് തടസ്സപ്പെടുത്തിയാല് ഓരോ കാലയളവിലും കടന്നുപോകുന്ന ജീനിനെ കണ്ടെത്താം. ജേക്കബ്, വുള്മാന് എന്നിവരാണ് ഈ രീതി ആവിഷ്കരിച്ചത്. ഇങ്ങനെ നിര്മിച്ച എസ്ചെരിഷ്യ കോളൈ ബാക്റ്റീരിയത്തിലെ വൃത്താകൃതിയായ ക്രോമസോമിന്റെ ഒരു മാപ്പ് ചിത്രത്തില് ചേര്ത്തിരിക്കുന്നു:
ബാക്റ്റീരിയ ജീന് മാപ്പിങ്ങിന്റെ മറ്റു മാര്ഗങ്ങള്. കോണ്ജുഗേഷനു പുറമേ ട്രാന്സ്ഡക്ഷന്, ട്രാന്സ്ഫൊര്മേഷന് എന്നീ മാര്ഗങ്ങളിലൂടെയും ബാക്റ്റീരിയങ്ങളില് ജീന് വിനിമയം നടക്കാം.
ഒരു ബാക്ടീരിയത്തില് നിന്നു മറ്റൊന്നിലേക്കു ചില ജീനുകള് ഒരു ബാക്റ്റീരിയാ വൈറസ് (bacterio phage) കൊണ്ടു പോകുന്നതാണു ട്രാന്സ്ഡക്ഷന്. ഒരുമിച്ച് കൊണ്ടുപോകപ്പെടുന്ന co -transduced)ജീനുകള് അടുത്തുള്ളവയാണെന്നു വ്യക്തം. ജീനുകളുടെ അകലം കുറഞ്ഞുവരുന്തോറും അവ ഒരുമിച്ചു കൊണ്ടുപോകപ്പെടുന്നതിന്റെ സാധ്യത കൂടും. ഈ ബന്ധം ചെറിയ അകലത്തിലുള്ള ജീനുകളെ മാപ്പു ചെയ്യാന് ഏറെ സഹായകമാണ്.
വളരുന്ന മാധ്യമത്തില് മറ്റു ബാക്റ്റീരിയങ്ങളില് നിന്ന് എത്തിയ ഡി.എന്.എ. ഖണ്ഡങ്ങളെ ഒരു ബാക്റ്റീരിയം എടുക്കുമ്പോഴുണ്ടാകുന്ന ജീന് വിനിമയമാണു ട്രാന്സ്ഫൊര്മേഷന്. ഇതില് ഒരുമിച്ച് ഉള്പ്പെടുന്ന ജീനുകള് അടുത്തടുത്തുള്ളവയായിരിക്കും.
വൈറസുകളുടെ ജീന് മാപ്പിങ്. ഒരു ബാക്റ്റീരിയാ വൈറസ് സ്പീഷീസിന്റെ ജനിതകമായി വ്യത്യസ്തമായ രണ്ടോ അതിലധികമോ ഇനങ്ങളെ ഒരുമിച്ച് ഒരു ബാക്റ്റീരിയത്തില് കടത്തിവിട്ടു നടത്തുന്ന പഠനത്തിലൂടെയാണു വൈറസുകളില് ജീന് മാപ്പിങ് നടത്തുന്നത്. ഒരേ ഡി.എന്.എ. തന്മാത്രതന്നെ ഒന്നിലധികം പ്രാവശ്യം ജീന് വിനിമയത്തില് ഉള്പ്പെടുക, ഒരു വൈറസിന്റെ ഒരു ഡി.എന്.എ. തന്മാത്രതന്നെ മറ്റു രണ്ടു വൈറസുകളുടെ വ്യത്യസ്ത ഡി.എന്.എ. തന്മാത്രകളുമായി ജീന് വിനിമയം നടത്തുക തുടങ്ങി പല സങ്കീര്ണതകളും വൈറസുകളുടെ ജീന്വിനിമയ മാപ്പിന്റെ നിര്മാണം ക്ളേശകരമാക്കുന്നു. എന്നാലും T2T4 തുടങ്ങി പല ബാക്റ്റീരിയോഫേജുകളുടെയും ജനിതക മാപ്പ് നിര്മിച്ചിട്ടുണ്ട്. പൊതുവെ അവ വൃത്താകൃതിയിലുള്ള ഡി.എന്.എ. തന്മാത്രകളെ പ്രതിനിധാനം ചെയ്യുന്നു.
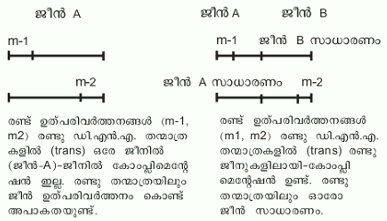
കോംപ്ലിമെന്റേഷന് മാപ്പിങ്. രണ്ടു ഡി.എന്.എ. തന്മാത്രകളിലായി കാണുന്ന രണ്ട് ഉത്പരിവര്ത്തനങ്ങള് (trans-mutations) ഒരേ ജീനില് തന്നെയാണെങ്കില് അവയ്ക്കു തമ്മില് പരസ്പരപൂരകമായി പ്രവര്ത്തിച്ച് (Complementation) സാധാരണ സ്വഭാവം (wild type) പ്രകടിപ്പിക്കാന് കഴിയില്ല. എന്നാല് രണ്ട് ഡി.എന്.എ. തന്മാത്രയിലുള്ള രണ്ട് ഉത്പരിവര്ത്തനങ്ങള് വ്യത്യസ്ത ജീനുകളിലാണെങ്കില് ഉത്പരിവര്ത്തനങ്ങളില്ലാത്ത രണ്ടു ജീനുകളും പരസ്പരപൂരകമായി പ്രവര്ത്തിച്ചു സാധാരണ സ്വഭാവം ഉണ്ടാക്കും.
ഈ രീതിയില് വ്യത്യസ്ത ജീനുകളെ തിരിച്ചറിയാനാകും. ഒരേ ജീനില്ത്തന്നെയുണ്ടാകുന്ന നിരവധി ഉത്പരിവര്ത്തനങ്ങള് തമ്മില് ജീന് വിനിമയം നടക്കാം. ഇതിനെ അടിസ്ഥാനപ്പെടുത്തി ഒരു ജീനിന്റെ തന്നെ സൂക്ഷ്മഘടന മാപ്പു ചെയ്യാന് കഴിയും.
ഡെലീഷിന് മാപ്പിങ്. ഒരു ജീനിന്റെ ഡി.എന്.എ.യില് വരുന്ന ന്യൂക്ളിയോറ്റൈഡ് വ്യത്യാസങ്ങളെ പോയിന്റ് ഉത്പരിവര്ത്തനം (point mutation) എന്നു പറയുന്നു. ഡി.എന്.എ.യുടെ കുറേ ഭാഗം നഷ്ടമാകുന്നതാണു ഡെലീഷന്. പോയിന്റ് ഉത്പരിവര്ത്തനങ്ങള് തമ്മില് ജീന് വിനിമയം നടക്കും. അതിന്റെ ഫലമായി സാധാരണ തരം (wild type) ജീന് ഉണ്ടാകാം. എന്നാല് ഒരു പോയിന്റ് ഉത്പരിവര്ത്തനവും അതുള്പ്പെടുന്ന ഡെലീഷനും തമ്മില് ജീന് വിനിമയം നടക്കില്ല. ഇതിനെ അടിസ്ഥാനമാക്കി നിരവധി പോയിന്റ് ഉത്പരിവര്ത്തനങ്ങള് ഏതു ഡെലീഷന് ഭാഗത്ത് ഉള്പ്പെടുന്നുവെന്ന് കണ്ടുപിടിക്കാം. ജീനിന്റെ സൂക്ഷ്മ ഘടന മാപ്പു ചെയ്യാന് ഈ രീതിയും സഹായകമാണ്.
മനുഷ്യന്റെ ജീന് മാപ്പിങ്. പരീക്ഷണങ്ങള്ക്കായി നിയന്ത്രിക്കാന് കഴിയാത്ത ഇണചേരലും കുട്ടികളുടെ കുറഞ്ഞ എണ്ണവും ഏറിയ ആയുര്ദൈര്ഘ്യവും മനുഷ്യന്റെ ജനിതക പഠനം വിഷമകരമാക്കുന്നു. തായ്വഴി പഠനം (pedigree analysis), സൊമാറ്റിക് സങ്കരണം (somatic hybridization), തന്മാത്രീയ മാര്ഗങ്ങള് (molecular mapping) എന്നിവയാണ് മനുഷ്യ ജീന് മാപ്പിങ്ങിന് ഉപയോഗിക്കുന്ന രീതികള്.
തായ്വഴി പഠനം. മനുഷ്യജനിതക പഠനത്തില് വിപുലമായി ഉപയോഗിക്കുന്നതാണ് തായ്വഴി പഠനം. ഇതില് ഒരു പ്രത്യേക സ്വഭാവം (trait), ഒരു കുടുംബത്തിലെ പല തലമുറകളിലെ വിവിധ വ്യക്തികളിലൂടെ എങ്ങനെ കടന്നു പോകുന്നുവെന്നു നിരീക്ഷിക്കുന്നു. മനുഷ്യന്റെ x ക്രോമസോമിലെ ജീനുകള് കാണിക്കുന്ന ക്രിസ്-ക്രോസ് പാരമ്പര്യം ആ ക്രോമസോമിലെ ജീനുകളെ കണ്ടെത്താന് സഹായിച്ചിട്ടുണ്ട്. അതു കൂടാതെ ക്രോമസോമുകളുടെ ഘടനാവൈകല്യങ്ങള് structural aberrations) സ്വഭാവങ്ങളില് വരുത്തുന്ന വ്യത്യാസങ്ങളെ നിരീക്ഷിച്ചും അവയുടെ ജീനുകളുടെ ക്രോമസോമിലെ സ്ഥാനം ഏകദേശം നിര്ണയിക്കാം.
സൊമാറ്റിക് സങ്കരണം. രണ്ടു ശരീരകോശങ്ങളെ തമ്മില് പോളി എത്തിലിന് ഗ്ലൈക്കോള് (PEG), വൈദ്യുതി തുടങ്ങിയ ഫ്യൂസജങ്ങളുടെ (fusogens) സഹായത്തോടെ ഒന്നിച്ചു ചേര്ക്കുന്ന രീതിയാണ് സൊമാറ്റിക് സങ്കരണം. ഏതു രണ്ടു കോശങ്ങളെയും ഇങ്ങനെ യോജിപ്പിക്കാം. ജനിതകമായ അടുപ്പം കുറെയൊക്കെ ഉള്ള രണ്ടു ചെടികളുടെ പ്രോട്ടോപ്ലാസ്റ്റുകളെ (protoplasts - കോശഭിത്തി മാറ്റിയ സസ്യകോശ) യോജിപ്പിച്ചാല് ഇതില് നിന്നു പുതിയ ഒരു സസ്യത്തെപ്പോലും ഉണ്ടാക്കാം. (ഉദാ. പൊട്ടറ്റോ + റ്റൊമാറ്റോ = പൊമാറ്റോ). എന്നാല് ജനിതക വൈജാത്യം ഏറെയുള്ള രണ്ടു ജീവികളുടെ കോശങ്ങളാണു യോജിപ്പിച്ചതെങ്കില് അതിന് ഒന്നിന്റെ ക്രോമസോമുകള് മാത്രം അടുത്ത ഓരോ കോശവിഭജനത്തിലും നഷ്ടപ്പെടുന്നതായി കണ്ടു വരുന്നു. മനുഷ്യന്റെയും എലിയുടെയും ശരീരകോശങ്ങള് തമ്മില് യോജിപ്പിച്ച് അതില് നിന്നു നഷ്ടപ്പെടുന്ന മനുഷ്യക്രോമസോമുകളെയും അവയിലെ ജീനുകളെയും പറ്റി പഠനങ്ങള് നടന്നിട്ടുണ്ട്. ഒരു പ്രത്യേക ജീനിനെയോ അതിന്റെ മെസഞ്ചര് ആര്.എന്.എ.യോ അടിസ്ഥാനമാക്കി നിര്മിച്ച ഒരന്വേഷകത്തെ (probe) ക്രോമസോമുമായി സ്വസ്ഥാന സങ്കരണം (in situ hybridization) നടത്തിയും ജീനിന്റെ ക്രോമസോമിലെ സ്ഥാനം കണ്ടെത്താം.
തന്മാത്രിക മാപ്പിങ്. ഒരു ജീവിയുടെ മൊത്തം ജീനുകളും അതിന്റെ കോശ ഡി.എന്.എ.യില് കാണുന്നു. ഇതിനെ ജീനോം (genome) എന്നു പറയുന്നു. ഏതെങ്കിലും ഉത്പരിവര്ത്തനം അറിയപ്പെട്ട ജീനുകളെ മാത്രമേ ജീന് വിനിമയ രീതിയില് മാപ്പു ചെയ്യാനാവു. ജീനോമിന്റെ മൊത്തം മാപ്പിങ്ങിന് ഇതു മതിയാവില്ല. ഇതിനായി തന്മാത്രിക അടയാളങ്ങള് (molecular markers) വളരെ സഹായകരമാണ്. ജീനോമിന്റെ മൊത്തത്തിലുള്ള ഒരു ഭൗതിക മാപ്പ് (physical map) നിര്മിക്കാന് ഈ രീതിയില് കഴിയും. ഇതിനു പല ജനറ്റിക് എന്ജിനീയറിങ് സങ്കേതങ്ങളും ഉപയോഗിക്കുന്നു.
റെസ്ട്രിക്ഷന് മാപ്പിങ്. ജീനോം ഡി.എന്.എ.യെ റെസ്ട്രിക്ഷന് എന്ഡോന്യൂക്ലിയേസ് എന്സൈമുകള് ഉപയോഗിച്ചു കഷണങ്ങളായി മുറിക്കുന്നു. ഈ കഷണങ്ങളെ അവയുടെ വലുപ്പമനുസരിച്ച് ഇലക്ട്രോഫോറസിസ് ഉപയോഗിച്ചു വേര്തിരിക്കുന്നു. കൃത്യമായ ഡി.എന്.എ. സീക്വന്സിലാണ് ഓരോ റെസ്ട്രിക്ഷന് എന്സൈമും ഡി.എന്.എ.യെ മുറിക്കുക. അതനുസരിച്ച് ഡി.എന്.എ. കഷണങ്ങളുടെ ക്രമം നിശ്ചയിക്കുന്നു. ഇതിനായി ക്രോമസോം നടത്ത (chromosome walking), ക്രോമസോം ചാട്ടം (chromosome jumping) എന്നീ രീതികളുണ്ട്. ഒരു ക്രോമസോമിലെ ഡി.എന്.എ.യുടെ റെസ്ട്രിക്ഷന് ഖണ്ഡങ്ങളുടെ ക്രമം കണ്ടെത്താനുള്ള രീതിയാണ് ക്രോമസോം നടത്ത. ഇത് ഡി.എന്.എ. ഖണ്ഡത്തില് നിന്നും തുടങ്ങുന്നു. ഇതിന്റെ അഗ്രഭാഗങ്ങളുമായി മേലേറ്റം (overlapping) ഉള്ള മറ്റു രണ്ടു ഖണ്ഡങ്ങളെ ഡി.എന്.എ. സങ്കരണത്തിലൂടെ (DNA hybridization) കണ്ടെത്തുന്നു. ഇങ്ങനെ ഖണ്ഡങ്ങളുടെ പൊതുവായ ഭാഗങ്ങളുടെ മേലേറ്റത്തെ അടിസ്ഥാനമാക്കി ക്രോമസോമിന്റെ ഒരു ഭാഗത്തു നിന്നും രണ്ടു ദിശയിലേക്കും നീങ്ങാം. ഒടുവില് മൊത്തം ക്രോമസോമിന്റെയും മാപ്പിങ് പൂര്ത്തിയാക്കാം. താരതമ്യേന വലിയ ഡി.എന്.എ. ഖണ്ഡങ്ങളുടെ ക്രോമസോമിലെ ക്രമനിര്ണയത്തിനാണ് ക്രോമസോം ചാട്ടം എന്നു പറയുന്നത്.
ഡി.എന്.എ. ക്രമനിര്ണയം (DNA sequencing). ഒരു ജീവിയുടെ എല്ലാ ജീനിന്റെയും (genome) മാപ്പിങ് പൂര്ണതയിലെത്തിക്കുവാന് ഡി.എന്.എ. ക്രമനിര്ണയം സഹായിക്കുന്നു. ഡി.എന്.എ. തന്മാത്ര നിരവധി നൂക്ലിയോറ്റൈഡുകള് കൊണ്ടാണ് നിര്മിച്ചിരിക്കുന്നത്. എന്നാല് നൂക്ലിയോറ്റൈഡുകള് നാലുതരം മാത്രമാണു ഡി.എന്.എ.യിലുള്ളത്. ഇവയുടെ ക്രമത്തിലെ വ്യത്യാസങ്ങളാണു പലതരം ജീനുകള് ഉണ്ടാകാന് കാരണം. ഡി.എന്.എ.യിലെ നൂക്ലിയോറ്റൈഡുകളുടെ ക്രമനിര്ണയത്തിനു രാസപരവും എന്സൈം നിയന്ത്രിതവുമായി മാര്ഗങ്ങളുണ്ട്. എ.എം. മാക്സം, ഡബ്ല്യു. ഗില്ബര്ട്ട് എന്നിവരാണ് രാസപരമായ രീതി കണ്ടെത്തിയത്. എഫ്. സാന്ഗര് എന്സൈം നിയന്ത്രിത രീതി രൂപപ്പെടുത്തി. രണ്ടു രീതിയിലും ഡി.എന്.എ.യുടെ ഒരു ഭാഗത്തു നിന്നും ഓരോ നൂക്ലിയോറ്റൈഡിനു മാത്രം നീളത്തില് വ്യത്യാസമുള്ള ഖണ്ഡങ്ങള് ഉണ്ടാക്കുകയാണ് ആദ്യം ചെയ്യുന്നത്. നാലുതരം നൂക്ലിയോറ്റൈഡുകളില് (A G T C) ഓരോന്നിനെയും കണ്ടെത്തി, ഡി.എന്.എ.യെ അവിടെ മുറിക്കാന് കഴിവുള്ള പ്രത്യേക രാസപദാര്ഥങ്ങള് ഉപയോഗിക്കുന്നു. അല്ലെങ്കില് എന്സൈം ഉപയോഗിച്ച് ഓരോ നൂക്ലിയോറ്റൈഡിന്റെ മാത്രം നീളവ്യത്യാസമുള്ള ഖണ്ഡങ്ങള് പകര്ത്തുന്നു. ഇത്തരം തുണ്ടുകളെ നാലുവരിയായി ഇലക്ട്രോ ഫോറസിസ് ഉപയോഗിച്ച് അവയുടെ നീളമനുസരിച്ച് വേര്തിരിക്കുന്നു. ഓരോ ന്യൂക്ലിയോറ്റൈഡിന്റെ മാത്രം നീളവ്യത്യാസമുള്ള ഈ ഖണ്ഡങ്ങളില് നിന്നും ഡി.എന്.എ.യുടെ ക്രമം കണ്ടെത്തുന്നു. അതിലുള്ള ജീനുകളെയും ജീന് ഉത്പന്നങ്ങളെയും അവ കാരണമുണ്ടാകുന്ന പ്രകടസ്വഭാവങ്ങളെയും ക്രമേണ കണ്ടെത്താനാവും. ഡി.എന്.എ. സീക്വന്സില് തുടങ്ങി സ്വഭാവത്തിലെത്തുന്ന ഇത്തരം പഠനങ്ങളാണ് 'റിവേഴ്സ് ജനറ്റിക്സ്'. സാധാരണ ജനിതക പഠനങ്ങള് (forward genetics) ഒരു സ്വഭാവത്തിന്റെ നിരീക്ഷണത്തില് തുടങ്ങി അവസാനം അതിന്റെ ജീനിനെ കണ്ടെത്തുകയാണ് ചെയ്യുന്നത്.
നിരവധി വൈറസുകളുടെയും ബാക്റ്റീരിയങ്ങളുടെയും ഡി.എന്.എ. സീക്വന്സ് കണ്ടെത്തിക്കഴിഞ്ഞു. മനുഷ്യന്റെ ഡി.എന്.എ.യിലുള്ള മുന്നൂറു കോടിയിലധികം നൂക്ലിയോറ്റൈഡുകളുടെ മൊത്തം സീക്വന്സ് കണ്ടെത്താനുള്ള സംരംഭമാണ് ഹ്യൂമന് ജീനോം പ്രോജക്ട്. നിരവധി രാഷ്ട്രങ്ങളിലെ പരശതം പരീക്ഷണ ശാലകളില് പരസഹസ്രം ശാസ്ത്രജ്ഞര് പങ്കെടുക്കുന്ന ഈ പ്രോജക്ട് ശാസ്ത്രചരിത്രത്തിലെ ഏറ്റവും ബൃഹത്തായ ഒരു പദ്ധതിയാണ്. 1980-കളില് തുടങ്ങിയ ഈ പദ്ധതി 2003-ഓടെ ഫലപ്രാപ്തിയിലെത്തുമെന്നാണ് കരുതുന്നത്. ഡി.എന്.എ. സീക്വന്സിലുള്ള എല്ലാ ജീനുകളെയും അവയുടെ പ്രകടസ്വഭാവങ്ങളെയും കണ്ടെത്താന് പിന്നെയും നാളേറെ വേണ്ടിവരും.
(പ്രൊഫ. കെ. രാജീവ്)